DeepMind的一款预测蛋白质3D结构的软件彻底改变了生物学。
撰文 | Ewen Callaway
翻译 | 施普林格·自然上海办公室
十多年来,分子生物学家Martin Beck和同事一直在尝试完成世界上最难的拼图之一:一个描绘人体细胞最大分子机器的高分辨率模型。
这个庞然大物名为核孔复合体(nuclear pore complex),它控制着分子从基因组所在细胞核的进进出出。每个细胞内都有数百个这种复合体,每个复合体由1000多个蛋白组成,这些蛋白在一个核膜孔的周围形成环状。
这1000多块拼图来自30多种以各种方式交织的蛋白基本单元。更复杂的是,对这些基本单元3D结构的实验解析综合了许多物种的结构,因此有时无法完美拼接。拼图盒子上的整体图——核孔复合体的低分辨率3D视图——并不够精细,无法判断其中有多少块拼图能完美拼合。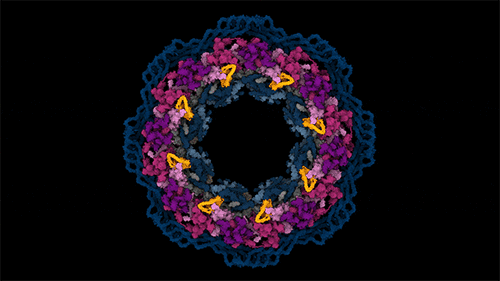 人核孔复合体的俯视图,核孔复合体是人体细胞中最大的分子机器。来源:Agnieszka Obarska-Kosinska
人核孔复合体的俯视图,核孔复合体是人体细胞中最大的分子机器。来源:Agnieszka Obarska-Kosinska
2016年,德国马克斯·普朗克生物物理研究所(MPIBP)的Beck领导团队报道了一个模型[1],这个模型名为Nup蛋白,覆盖了约30%的核孔复合体,占到了这30多个基本单元的约一半。
去年7月,谷歌母公司Alphabet位于伦敦的子公司DeepMind公开了名为AlphaFold的AI工具[2](参见:全民可用的蛋白质结构预测来了!AlphaFold2源代码免费公开)。该软件能根据蛋白质的基因序列预测其3D结构,且大部分情况下准确度极高。这完全颠覆了Beck的工作,也彻底改变了成千上万生物学家的研究(见“AlphaFold狂热”)。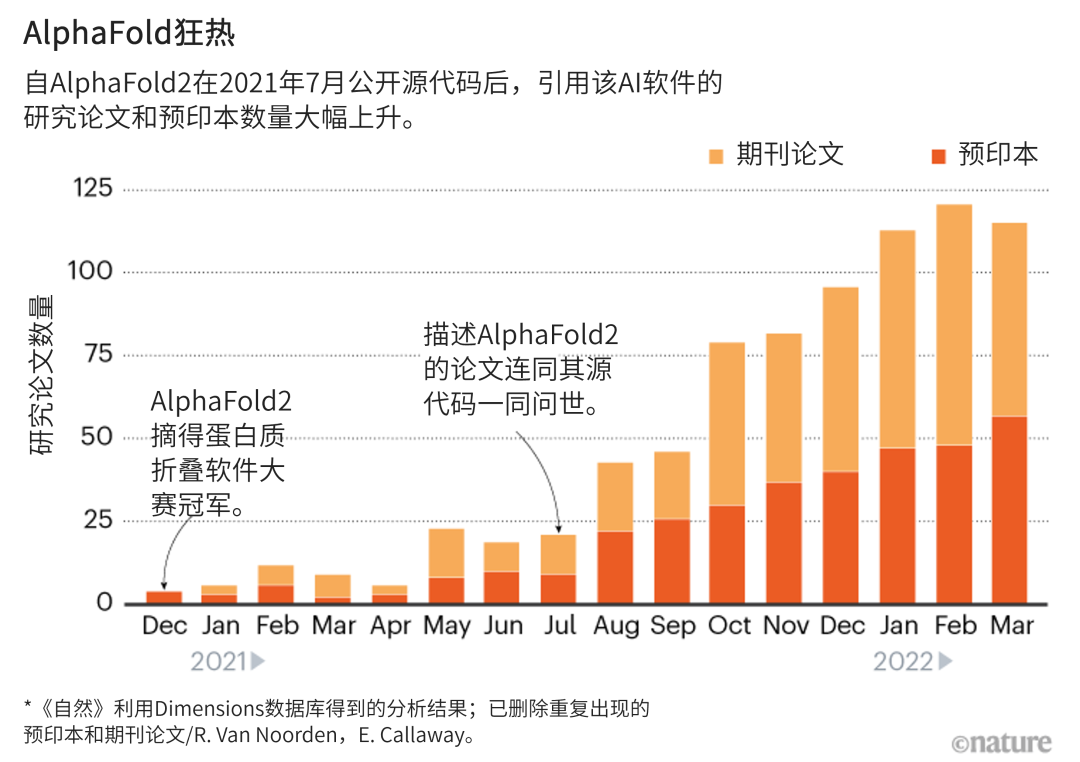 Beck说:“AlphaFold改变了整个游戏。”希伯来大学计算结构生物学家Ora Schueler-Furman说:“就像发生了一次地震。现在处处都能看到AlphaFold的身影。”Schueler-Furman使用AlphaFold模拟蛋白的相互作用。“世界分成了7月前的世界和7月后的世界。”
Beck说:“AlphaFold改变了整个游戏。”希伯来大学计算结构生物学家Ora Schueler-Furman说:“就像发生了一次地震。现在处处都能看到AlphaFold的身影。”Schueler-Furman使用AlphaFold模拟蛋白的相互作用。“世界分成了7月前的世界和7月后的世界。”
Beck和MPIBP的同事——分子生物学家Agnieszka Obarska-Kosinska和生物物理学家Gerhard Hummer领导的团队——以及欧洲分子学生物实验室(EMBL)的结构建模师Jan Kosinski带领的团队利用AlphaFold更准确地预测了人体Nup蛋白的结构。通过一个能让AlphaFold模拟蛋白相互作用的微小调整,他们在去年10月发表了一个能覆盖60%的核孔复合体的模型[3]。这个模型揭示了该复合体如何稳定细胞核的孔,同时提示了这些复合体以何种方式控制进进出出的分子。
过去半年里,生命科学领域掀起了一股AlphaFold狂热。伦敦大学学院的计算生物学家Christine Orengo说:“我参与的每个会议上,人们都在问‘干嘛不用AlphaFold?’。” 有时候,这个AI能节省科学家的大量时间;有时候,它能让之前无法想象或不切实际的研究成为可能。但它也存在局限性,有些科学家在自己的工作中发现它的预测不太可靠。但无论如何,这场实验正以疯狂的速度进行着。
即使是该软件的开发者,也在努力跟上它在从药物发现到蛋白设计再到复杂生命起源的各种应用。“我早上起来在Twitter里输入AlphaFold,”DeepMind的AlphaFold团队负责人John Jumper说,“看到屏幕上的一切,这种体验非常特别。”
一鸣惊人
AlphaFold在2020年12月曾引起轰动(参见:颠覆生物学!AlphaFold成功预测蛋白质结构)。当时,它在名为蛋白质结构预测关键评估(CASP)的大赛上一举夺魁。该比赛每两年举办一次,呈现了生物学界在一项艰巨挑战上的最新进展,这项挑战是如何仅凭蛋白质的氨基酸序列确定其3D结构。计算机软件给出的结构会拿来与经过X射线晶体学或冷冻电镜(cryo-EM)这类实验方法确认的结果进行对比。X射线晶体学和冷冻电镜分别通过X射线和电子束照射蛋白质的方式来对蛋白质的结构进行成像。
2020版的AlphaFold是该软件的第二个版本。AlphaFold之前还摘得了2018年CASP大赛的冠军(参见:DeepMind的Al不仅能打《星际争霸》,AlphaFold还能解析蛋白结构),但它的更早版本并不够好,不能取代使用实验方法解析的结构,Jumper说。不过,AlphaFold2的预测结果平均而言已与实验结果相差无几。
之前大家并不知道DeepMind何时会全面开放这个软件或它的预测结果,所以有研究人员利用Jumper在一次公开演讲中的信息以及他们自己的智慧开发了另一个AI工具,名为RoseTTAFold。
到了2021年7月15日,描述RoseTTAFold和AlphaFold2的论文相继发表[2, 4],同时公开的还有免费的开源代码等信息,让专业人士可以打造属于自己的版本。一周后,DeepMind宣布已经用AlphaFold预测了人体内近乎所有蛋白质的结构,以及20个其他被大量研究的生物体的完整“蛋白质组”,其中包括小鼠和大肠杆菌,累计共有36.5万个结构(见“已知的蛋白质组”;参见:颠覆生命科学!AlphaFold预测完整人类蛋白质组结构)。DeepMind还将这些信息上传到了由EMBL欧洲生物信息学研究所(EMBL–EBI)维护的数据库。在那之后,这个数据库已经收录了近100万个结构。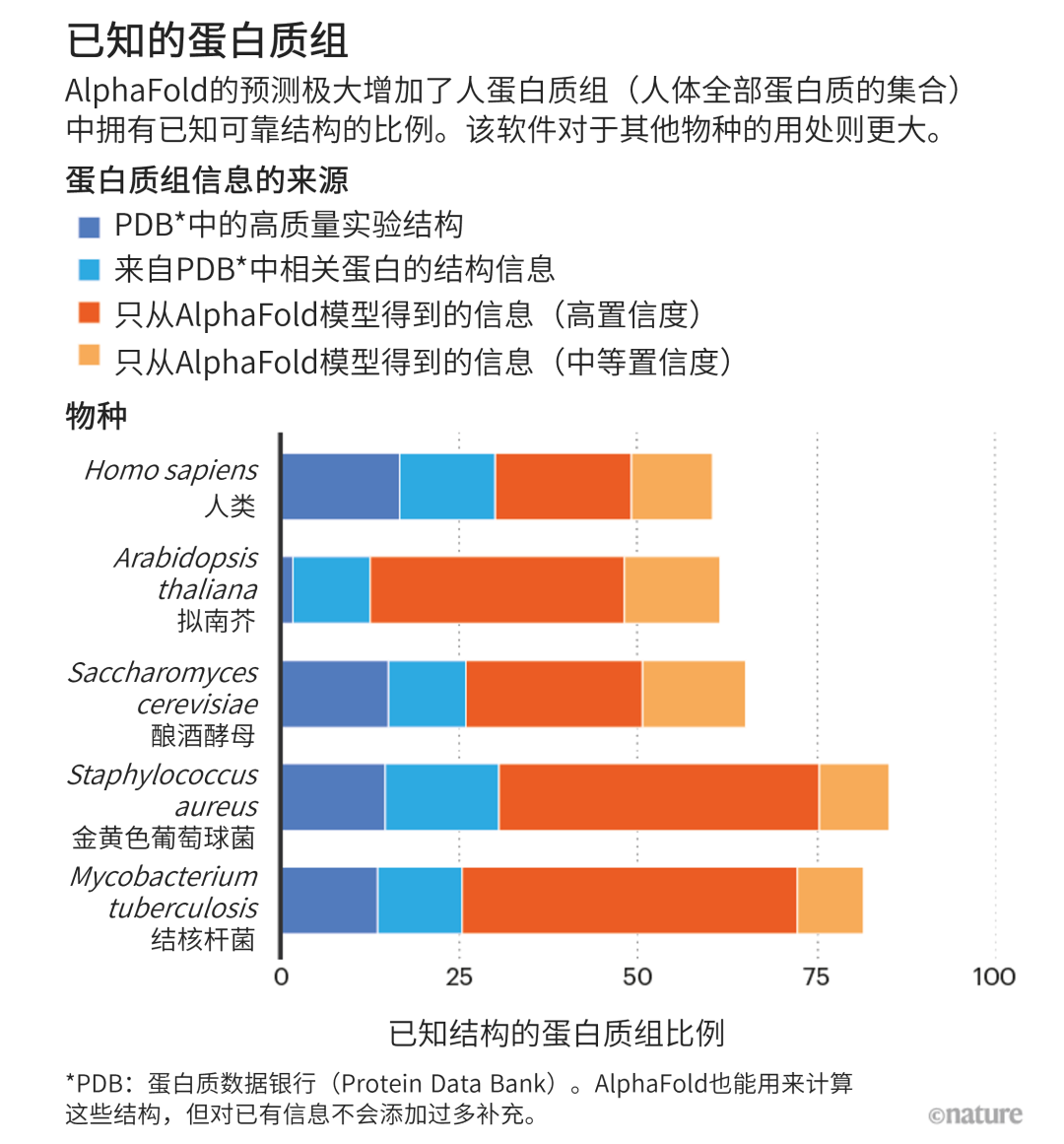 来源:E. Porta-Pardo et al. PLoS Comput. Biol. 18, e1009818 (2022).
来源:E. Porta-Pardo et al. PLoS Comput. Biol. 18, e1009818 (2022).
今年,DeepMind计划发布总计1亿多个结构预测——相当于所有已知蛋白的近一半,是蛋白质数据银行(PDB)结构数据库中经过实验解析的蛋白数量的几百倍之多。
AlphaFold使用了深度学习神经网络:受到大脑神经网络启发的计算机架构,能识别数据中的模式。该网络使用了上万个经过实验测定的蛋白质结构以及PDB等数据库中的序列进行训练。如果给它一个新序列,它会先在数据库中寻找类似序列,发现容易发生共同演化的氨基酸,提示它们的3D结构也很相似。已有的相关蛋白质结构为估算新序列中氨基酸对的距离提供了另一种方式。
在尝试模拟氨基酸的3D构象时,AlphaFold会反复迭代这些并行跟踪的线索,并持续更新它的最新预测。专业人士表示,AlphaFold对于机器学习领域最新概念的应用似乎是让它出类拔萃的关键——尤其是它使用的一个名为“attention”的AI机制来确定哪些氨基酸连接方式在当前任务中是最突出的。
由于该网络需要依赖相关蛋白序列的信息,这也意味着AlphaFold存在一定的局限性。AlphaFold无法用来预测突变对蛋白质结构的影响——比如那些会致病的突变。它也无法通过训练来确定蛋白质在遇到其他相互作用蛋白或药物等分子的情况下会如何变化。但它的模型能为它对蛋白质各氨基酸单元的预测进行置信度打分——研究人员一直在修改AlphaFold的代码,以便进一步提升其能力。
根据DeepMind的统计,目前已有超40万人使用过EMBL-EBI的AlphaFold数据库。此外还有一些AlphaFold的“超级用户”:这些研究人员在自己服务器上安装了AlphaFold,或是打造了AlphaFold的云版本,用来预测不在EMBL-EBI数据库中的结构,或是探索AlphaFold的新用途。
解析结构
生物学家对AlphaFold的结构解析能力叹为观止。“基于我目前看到的结果,我很信任AlphaFold。”丹麦奥尔胡斯大学的结构生物学家Thomas Boesen说道。这个软件成功预测了Boesen所在机构已经测定但尚未发表的蛋白质结构。他说:“这是来自我们的一次极大的肯定。”他和奥尔胡斯大学的微生物生态学家Tina Šantl-Temkiv正在使用AlphaFold模拟能促进冰形成的细菌蛋白的结构,这种结构或许让云层中的冰具有降温效应,生物学家之前一直没能完全用实验方法解析这种结构[5]。
只要一种蛋白质能折叠形成单一、清晰的3D结构——并非所有蛋白质都会这样——AlphaFold的预测就基本不会输,斯德哥尔摩大学的蛋白质生物信息学家Arne Elofsson说,“只要点点鼠标,你大概就得到了最好的模型。”
Elofsson说,在AlphaFold置信度欠佳的领域,“它也能清楚地告诉你它对哪些预测是没把握的”。这时,它给出的预测结构有点像漂浮的意大利面(见“好的、坏的、糟糕的”)。这常出现在缺乏清晰结构的蛋白质区域——至少是在独立的区域。在这些固有无序的区域大概占到人体蛋白质组的1/3,可能只有在遇到另一个分子,如信号转导配偶体时,其结构才会变得清晰。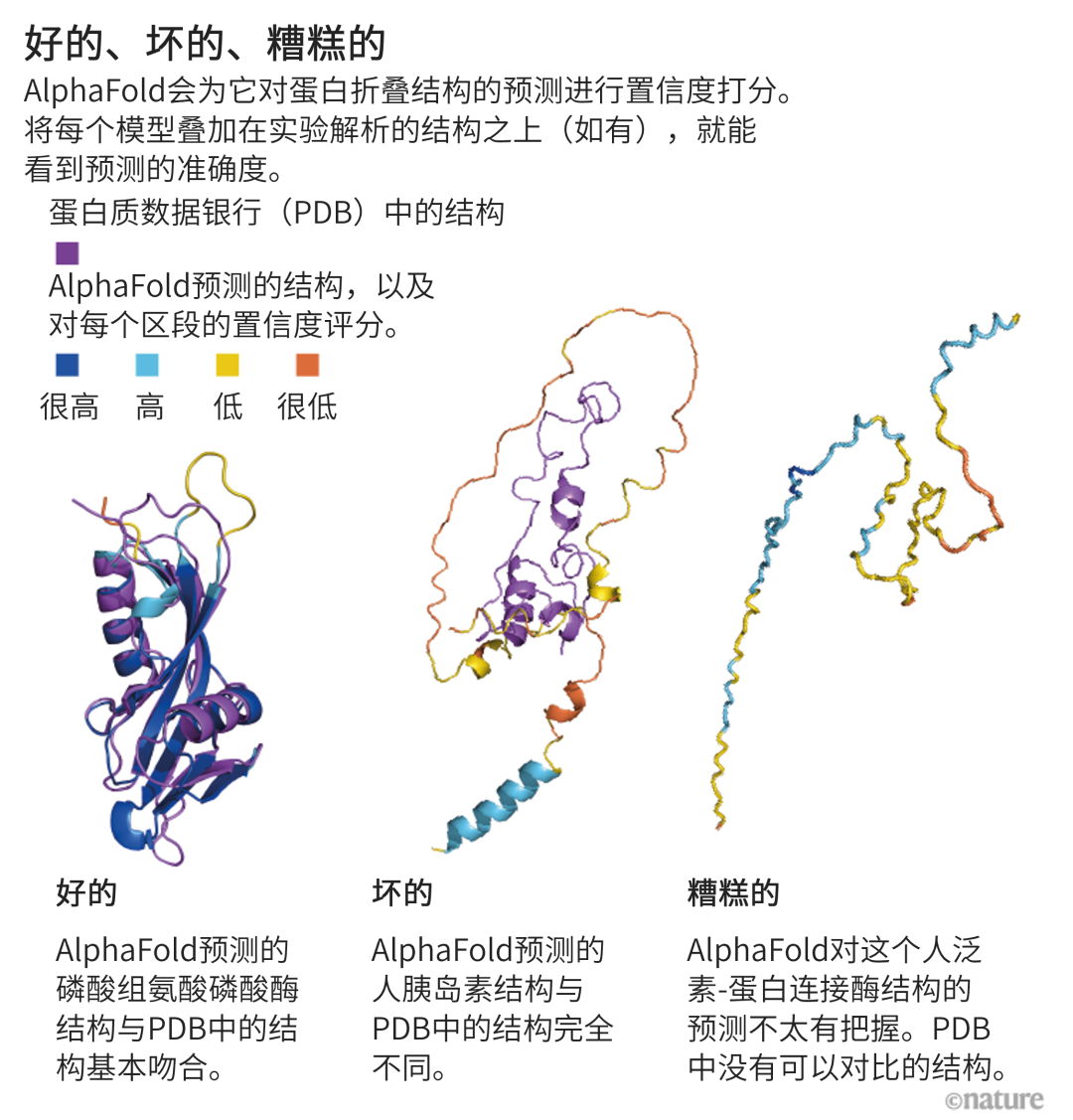
来源:J. M. Thornton et al. Nature Med. 27, 1666–1669 (2021).
英国癌症研究院的计算生物学家Norman Davey认为,AlphaFold识别无序结构的能力彻底改变了他对这些区域性质的研究。他说:“我们的预测质量一夜之间突飞猛进,而我们什么都没做。”
AlphaFold在EMBL-EBI数据库上传的大量蛋白质结构也立即得到了使用。Orengo的团队正在通过搜索数据库发现新的蛋白质类型(未经过实验验证),并已经发现了数百乃至数千个潜在的新蛋白家族,扩充了科学家对蛋白质形状和功能的认知。该团队还在搜索另一个包含从海洋和废水中提取的DNA序列的数据库,试图发现新的食塑酶。通过让AlphaFold快速预测数千个蛋白的结构,该团队希望能更好地理解酶是如何通过演化拥有分解塑料的能力,并能进一步优化它们。
哈佛大学演化生物学家Sergey Ovchinnikov认为,这种将任何蛋白编码基因序列转化为可靠结构的能力,对于演化研究来说非常有用。研究人员通过比较基因序列来确定不同物种的生物体与它们基因之间的亲缘关系。对于亲缘关系较远的基因来说,这种比较可能无法发现演化近亲,因为这些序列已经变了很多。但通过比较变化速度比基因序列更慢的蛋白质结构,研究人员或许能揭示之前没注意到的古代关系。苏黎世联邦理工学院的计算生物学家Pedro Beltrao说:“这为蛋白质演化和生命起源的研究带来了难能可贵的机会。”
为测试这一概念,首尔大学计算生物学家Martin Steinegger领导的一个团队和他的同事利用他们开发的工具Foldseek,在EMBL-EBI的AlphaFold数据库中寻找新冠病毒(SARS-CoV-2)RNA复制酶的近亲[6],结果找到了之前未被发现的可能古代近亲:真核生物(包括黏菌类)蛋白的3D结构很像HIV等病毒用来将RNA复制成DNA的反转录酶,尽管两者在遗传序列水平上的相似度很低。
实验辅助
对于想要得到特定蛋白精细结构的科学家,AlphaFold的预测并非最直接的解决方案。确切地说,AlphaFold能提供一个初步预测,之后再由实验验证或优化。比如,来自X射线晶体学的原始数据以衍射X射线的图样呈现。一般来说,科学家需要对蛋白质结构有个初步猜测,才能解释这些图样。之前,他们通常需要拼凑PDB中相关蛋白的信息,或是利用实验方法,英国剑桥大学的结构生物学家Randy Read说。Read的实验室很擅长其中一些方法。如今,Read说,AlphaFold的预测让大部分X射线图样不再需要这些方法,他的实验室也尝试在实验模型中更好地运用AlphaFold。“我们的研究重点完全变了。”
他和其他研究人员已经通过AlphaFold利用X射线数据确定了晶体结构,这些数据本来在没有适当初始模型的前提下是无法解读的。“现在大家可以解析几年来都无法解析的结构了。”Read实验室的前博士后研究员、如今就职于分析公司SciBite的Claudia Millán Nebot表示。她预计将有大量新的蛋白质结构上传到PDB,而且绝大部分都来自AlphaFold。
专门使用冷冻电镜的实验室也是同样的情况——冷冻电镜能拍下急速冷冻的蛋白照片。有时,在其他软件会搞错的情况下,AlphaFold的模型却能准确预测重要的药物靶标——G蛋白偶联受体(GPCR)的独有特征,北卡罗来纳大学教堂山分校的结构生物学家兼药理学家Bryan Roth说,“它似乎能给出很好的初始模型,之后可以再用一些实验数据进行优化,”他说,“这替我们省了不少时间。”
但Roth也指出,AlphaFold并不是万无一失。在他们实验室已经解析出来但尚未发表的几十个GPCR结构中,“约有一半的情况下,AlphaFold预测的结构没有问题,但也有一半的情况下,AlphaFold的结构对于我们的目标没什么帮助”。他说,有时AlphaFold对于它的预测给出了很高的置信度,但实验结构显示它是错的。即使AlphaFold搞对了,它也无法模拟某个蛋白与药物或其他小分子(配体)结合时的样子,而这种情况会完全改变整个结构。这类注意事项让Roth不禁思考AlphaFold对于药物发现的作用究竟有多大。
药物发现领域正在越来越多地使用计算机对接软件从数十亿计的小分子中筛选能与蛋白结合的小分子——这个指标提示它们能成为真正的药物。Roth目前正在与加州大学旧金山分校的药物化学家Brian Shoichet合作,看看这种情况下AlphaFold的预测结果与实验解析的结构有何差异。
Shoichet表示,他们现在只研究AlphaFold的预测结果与实验结构相符的蛋白。但即使对于这些蛋白来说,对接软件对于实验结构和AlphaFold的结构也会给出不同的候选药物,这说明差异再小也有影响。Shoichet说:“这不是说我们找不到新的配体了,而是说我们会发现不一样的配体。”他的团队正在合成利用AlphaFold结构识别出的潜在药物,并在实验室检测这些药物的活性。
批判乐观
药企和生物科技公司的研究人员对于AlphaFold助力药物发现的潜力感到很兴奋,Shoichet说,“我会形容为批判乐观。”2021年11月,DeepMind成立了它自己的衍生公司——Isomorphic Labs。这家公司致力于将AlphaFold一类的AI工具应用到药物发现中。但该公司并未过多透露其他计划。
总部位于纽约的药物发现公司Schrödinger也专门开发化学模拟软件,该公司治疗研发主管Karen Akinsanya表示,她和她的同事利用AlphaFold预测的结构(包括GPCR的结构)在候选药物的虚拟筛选以及化合物设计上已经小有进展。她发现,和实验解析的结构一样,仍需另外的软件来确定氨基酸侧链的具体细节或个别氢原子的位置。但只要完成了这一步,AlphaFold预测的结构在某些情况下就能用来指导药物发现。
Akinsanya说:“很难说‘这就是万能之计’;就好像你把某个结构搞得很清楚,异常清楚,它就可以适用于所有结构了。但事实显然不是这样的。”她和同事已经发现AlphaFold对预测准确度的评分并不显示某个结构对之后的药物筛选是否有用。她说,对于药物研发来说,AlphaFold的预测永远无法取代实验解析的结构。但它们或许可以对实验方法进行补充,加快研究进度。
对AlphaFold跃跃欲试的药物研发机构在1月听到了好消息,当时,DeepMind决定清除AlphaFold实现商业应用的一个主要障碍。当DeepMind在2021年7月公开AlphaFold的代码时,它曾要求运行AlphaFold神经网络所需的参数或称权重——即利用数十万蛋白质结构和序列训练该网络的最终结果——只限非商业使用。Akinsanya说,这对行业内的一些机构来说是个瓶颈,而当DeepMind转变态度时,大家“激动极了”。(RoseTTAFold也有类似的限制,其中一位开发者Ovchinnikov说。但下一个版本将是完全开源的。)
AI工具不仅改变了科学家测定蛋白质结构的方式。一些研究人员还在利用这些工具打造全新的蛋白质。“深度学习彻底改变了我们团队设计蛋白质的方式。”华盛顿大学生物化学家、蛋白质设计和结构预测领域带头人David Baker说。Baker的团队和计算机化学家Minkyung Baek一起主导了RoseTTAFold的开发工作。
Baker的团队让AlphaFold和RoseTTAFold来“幻想”新的蛋白。他们改写了该AI的代码,让软件在得到随机氨基酸序列的情况下,对它们进行优化,直到合成出能被这些神经网络识别为蛋白的东西(见“幻想蛋白”)。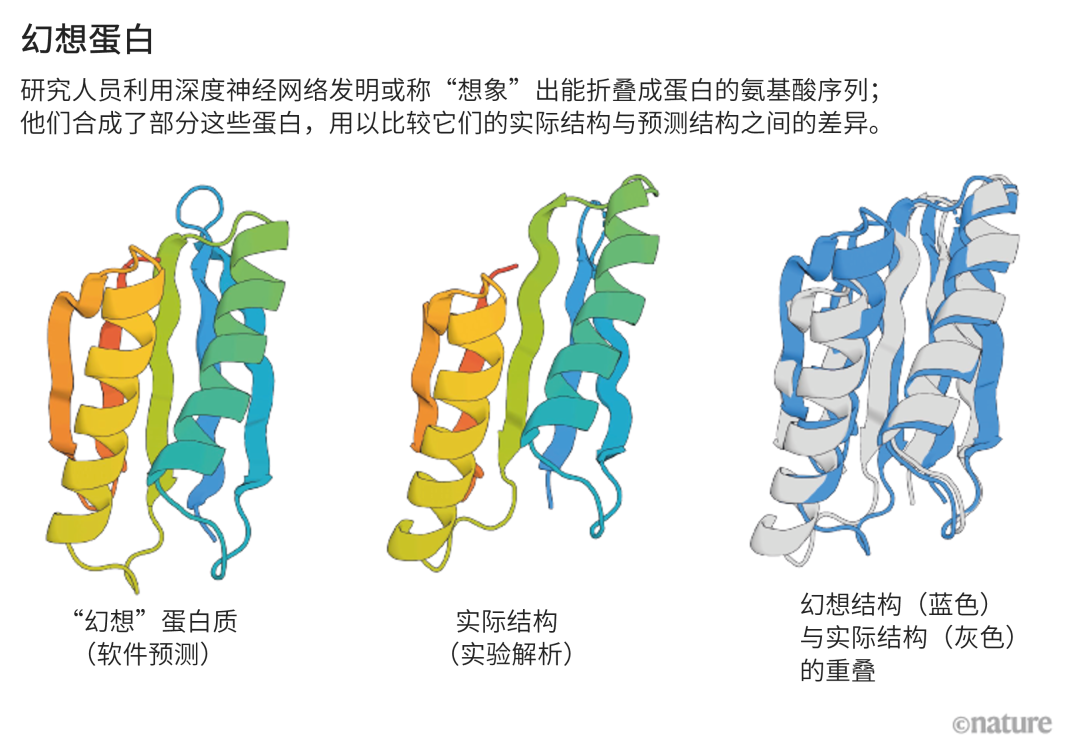 来源:参考文献[7]
来源:参考文献[7]
2021年12月,Baker和他的同事报道了他们在细菌中表达了129种这些幻想蛋白,发现其中约1/5的蛋白会折叠成类似他们预测的结构[7]。Baker说:“这是这种网络能用来设计蛋白质的首个证明。”他的团队现在正在利用这种方法设计有实际用途的蛋白质,比如能催化某类化学反应的蛋白质,方法是明确发挥目标功能的氨基酸并把接下来的工作都交给AI。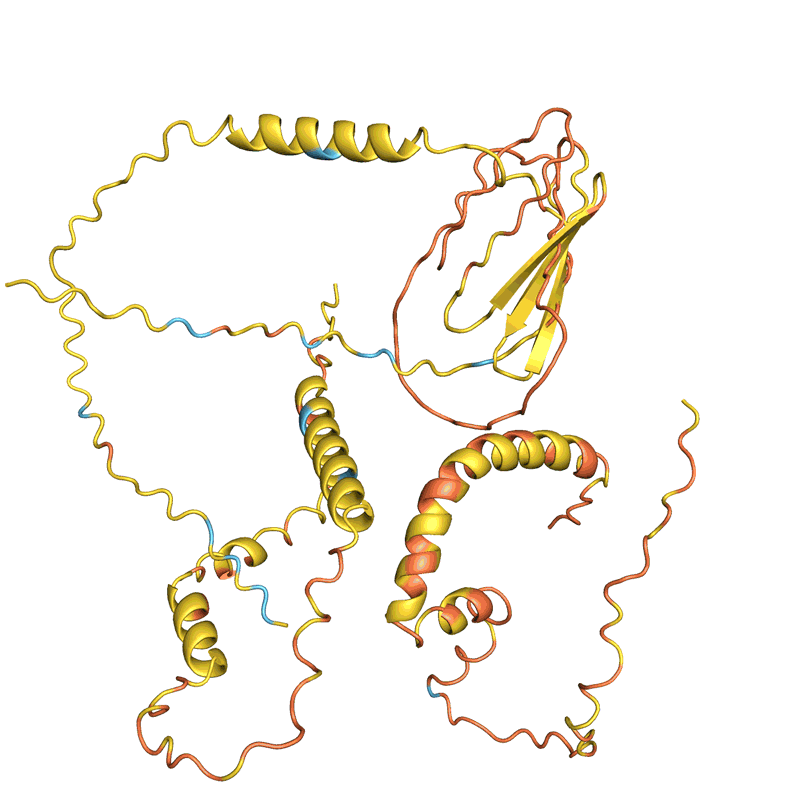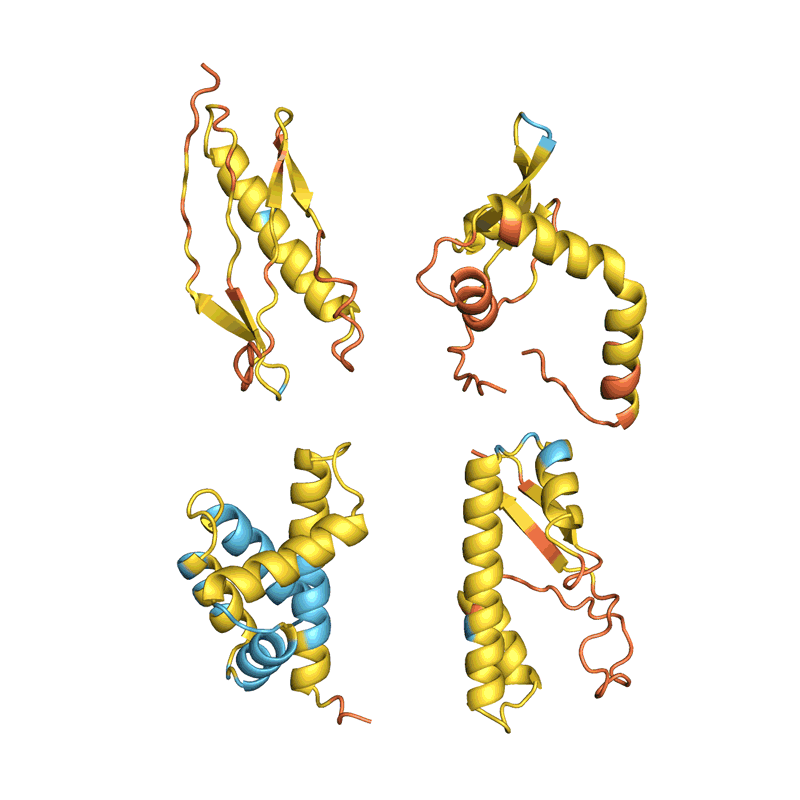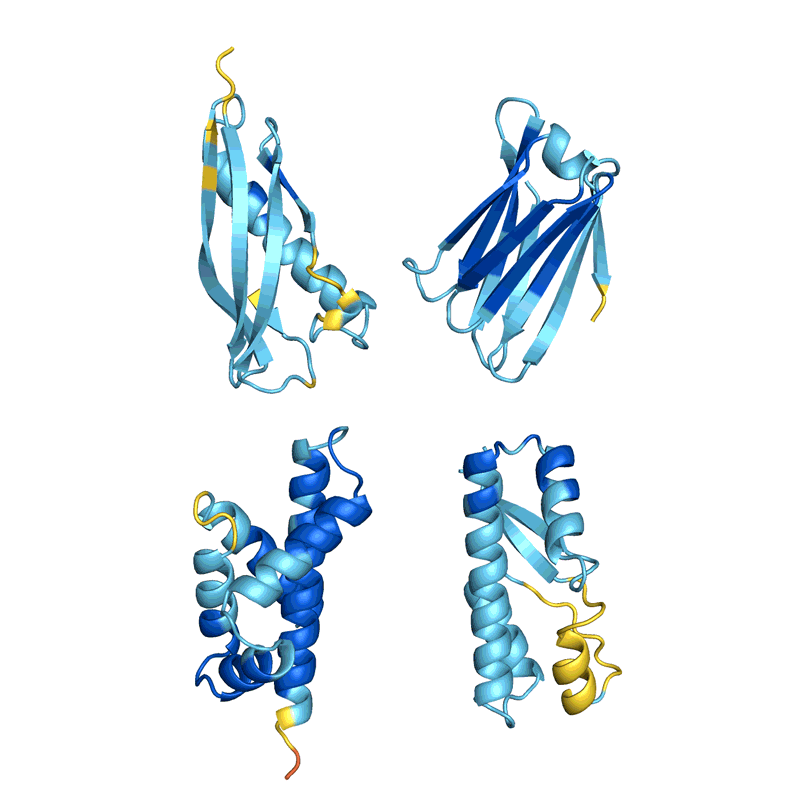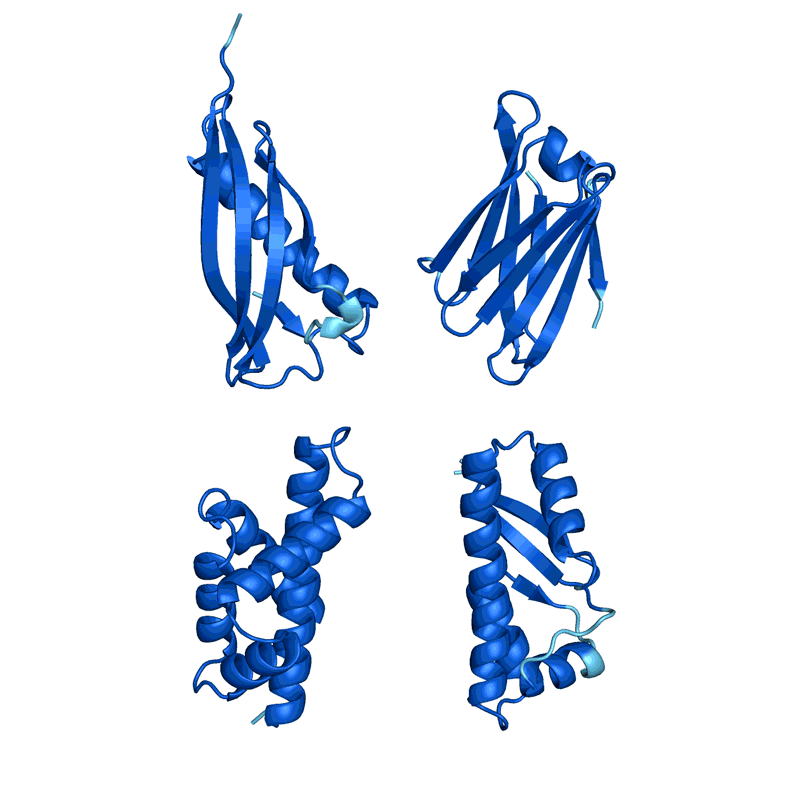 蛋白“幻想”的四个例子。每一例中,AlphaFold都会收到一个随机的氨基酸序列,它会预测其结构,改变序列,直到AlphaFold有把握预测它将折叠成具有清晰3D结构的蛋白质。颜色代表预测置信度(从极低到极高分别为红色、黄色、浅蓝色、深蓝色)。为清晰起见,原始速度被减慢。来源:Sergey Ovchinnikov
蛋白“幻想”的四个例子。每一例中,AlphaFold都会收到一个随机的氨基酸序列,它会预测其结构,改变序列,直到AlphaFold有把握预测它将折叠成具有清晰3D结构的蛋白质。颜色代表预测置信度(从极低到极高分别为红色、黄色、浅蓝色、深蓝色)。为清晰起见,原始速度被减慢。来源:Sergey Ovchinnikov
黑客攻击
当DeepMind刚公布AlphaFold的代码时,Ovchinnikov很想搞清楚它是如何进行预测的。于是,他和计算生物学的同事们,包括Steinegger在内,用了几天时间创建了一个名为ColabFold的网站。该网站能让所有人向AlphaFold或RoseTTAFold上传蛋白质序列,并得到预测结果。Ovchinnikov起初的设想是,通过提供关于目标蛋白序列演化近亲的错误信息,他们能用ColabFold尝试“破解”AlphaFold。Ovchinnikov希望能用这种方式弄明白这个网络的预测精度为何这么高。
结果,大部分使用ColabFold的研究人员只想获得某个蛋白质的结构。但也有人将它作为一个平台,修改输入AlphaFold的内容,探索新的用途。Jumper说:“我没想到有那么多不同类型的黑客攻击手段。”
目前为止,最常用的攻击手段是将其用在多个相互作用且互相缠绕的肽链组成的蛋白复合物上。和核孔复合体一样,细胞内许多蛋白质都是在与多个蛋白亚基形成复合物的同时获得功能。
AlphaFold的设计目标旨在预测单个肽链的结构,对它的训练也全由这类蛋白质组成。但这个网络似乎对复合体的折叠方式也略知一二。就在AlphaFold代码公开的几天后,东京大学的生物信息学家森胁由隆(Yoshitaka Moriwaki)发推特说,AlphaFold能准确预测用一个长连接序列连接的两个蛋白序列之间的相互作用。Baek很快也分享了在开发RoseTTAFold过程中发现的另一种预测复合物的攻击办法。
ColabFold之后也加入预测复合物的能力。2021年10月,DeepMind发布了名为AlphaFold-Multimer的更新[8]——和之前的版本不一样,这个是专门使用蛋白质复合物进行训练的。Jumper的团队将其应用于PDB中的数千个复合物上,发现它能预测约70%的已知蛋白-蛋白相互作用。
研究人员已经在用这些工具发现潜在的新蛋白配对。Elofsson的团队利用AlphaFold预测了6.5万个人体蛋白对的结构——实验数据显示这些蛋白对会相互作用[9]。Baker领导的一个团队用AlphaFold和RoseTTAFold模拟了酵母编码的近乎每对蛋白之间的相互作用,发现了100多种之前未知的复合物[10]。这类筛选工作还只是起步,Elofsson说。它们在预测某些蛋白质配对方面的能力很强,尤其是那些很稳定的蛋白质对,但它们发现短暂互作的能力略差。“看起来好不等于就是正确的,”Elofsson说,“你需要实验数据支持你是对的。”
Kosinski认为,核孔蛋白复合体的例子很好地体现了预测数据可以和实验数据相结合(见“基因组通道”)。“这不是说我们把所有30个蛋白都扔给AlphaFold去预测结构。”为了把预测好的蛋白质结构拼凑起来,团队利用名为冷冻电子断层的冷冻电镜技术获得核孔复合物3D图像(参见:打开全新“视”界:冷冻电镜成像技术首达原子分辨率)。在一例中,能测定蛋白质之间距离的实验发现了该复合物两个成分之间令人意外的相互作用,并得到了AlphaFold模型的确认。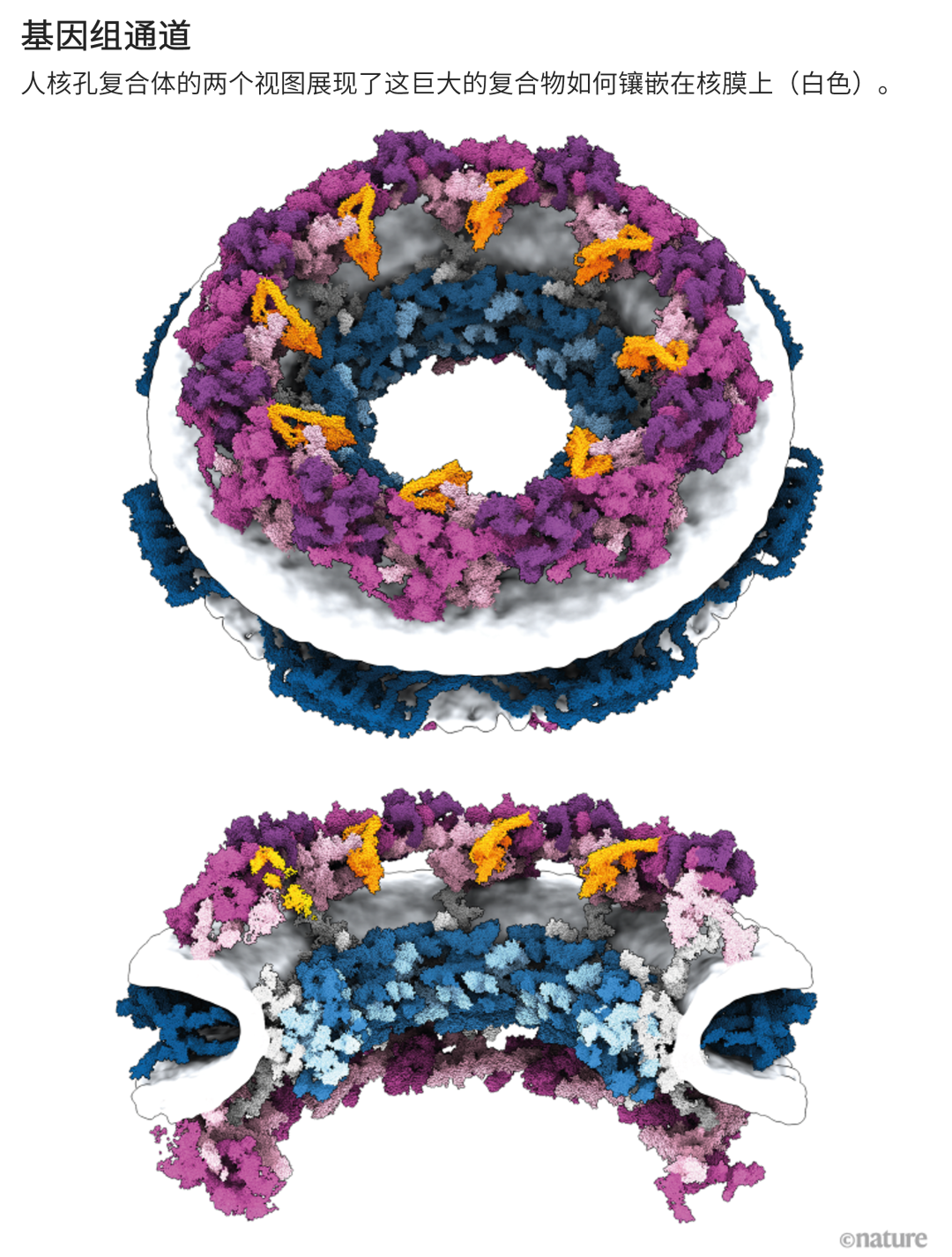 图片改编自参考文献[3]/Agnieszka Obarska-Kosinska
图片改编自参考文献[3]/Agnieszka Obarska-Kosinska
Kosinski团队将他们得到的核孔复合物的最新图谱比作一个起点,后续将有进一步实验和模拟来研究这些核孔复合物的功能,以及它如何在疾病中丧失功能。
AlphaFold的局限
虽然AlphaFold的出现推动了巨大进展,但科学家认为有必要说明它的局限性——尤其是因为非蛋白质预测专业的研究人员也会用到它。
一些研究人员尝试将AlphaFold应用于会破坏蛋白天然结构的各类突变,包括与早期乳腺癌有关的一个突变,这些尝试证实了AlphaFold还无法预测新的突变对蛋白质的影响,因为没有演化上相关的序列可以用来研究[11]。
AlphaFold团队正在思索如何设计出针对新突变的神经网络。为此,Jumper认为该网络需要能更好地预测蛋白如何从未折叠状态变成折叠状态。而这需要软件仅凭对蛋白质物理特性的了解就能预测结构,纽约哥伦比亚大学计算生物学家Mohammed AlQuraishi说。“我们的一个设想是在不借助演化信息的情况下基于单个序列展开预测,”他说,“这是仍有待解决的一个关键问题。”
AlphaFold也可以用于预测单个结构,虽然研究人员已经通过攻击训练它给出不止一个结构。但是,许多蛋白质有多个构象,这些构象可能对它们的功能很重要。Schueler-Furman说:“AlphaFold无法很好地处理会在不同构象中呈现不同结构的蛋白。”这些预测是针对单独的结构,而许多蛋白质其实会与配体一起发挥功能,如DNA和RNA、脂肪分子、铁等矿物质。Elofsson说:“我们还是缺少配体,我们缺少关于蛋白质的其他一切。”
AlQuraishi认为,下一代神经网络的开发面临着巨大的挑战。AlphaFold的背后是数十年的研究成果,将这些实验测得的结构拿来学习。对于蛋白质动力学,或是能与蛋白质相互作用的无数小分子的结构来说,并没有这个体量的数据可以推动进展。Jumper说,PDB 包含了蛋白质在与其他分子相互作用时的结构,但这只代表了化学多样性的一小部分。
研究人员认为,他们还要过一段时间才能弄明白AlphaFold这类AI工具的最佳用法。AlQuraishi形容这好比电视机刚出现的时候,一些电视节目只是在用广播报新闻。“我相信今后我们会找到当前还无法想象的新应用。”
AlphaFold掀起的革命会走向何方无法预见。“变化的速度太快了,”Baker说,“即使在下一年,我们还将看到这些工具推动的巨大突破。”EMBL-EBI的计算生物学家Janet Thornton认为AlphaFold带来的最大转变之一,可能是让生物学家更愿意接受计算机和理论的研究方法。她说:“对我来说,真正的变革是人们思维方式的变化。”
AlphaFold革命让Kosinski可以大胆畅想未来。他想象以AlphaFold为灵感的工具今后不仅能用来模拟单个蛋白和复合物,还能模拟整个细胞器,甚至是在单个蛋白分子水平上的细胞。“这是我们将在接下来的几十年里追逐的梦想。
参考文献
[1] Kosinski, J. et al. Science 5, 363–365 (2016).
[2] Jumper, J. et al. Nature 596, 583–589 (2021).
[3] Mosalaganti, S. et al. Preprint at bioRxiv https://doi.org/10.1101/2021.10.26.465776 (2021).
[4] Baek, M. et al. Science373, 871–876 (2021).[5] Hartmann, S. et al. Preprint at bioRxiv https://doi.org/10.1101/2022.01.21.477219 (2022).
[6] van Kempen, M. et al. Preprint at bioRxiv https://doi.org/10.1101/2022.02.07.479398 (2022).[7] Anishchenko, I. et al. Nature600, 547–552 (2021).[8] Evans, R. et al. Preprint at bioRxiv https://doi.org/10.1101/2021.10.04.463034 (2021).
[9] Bryant, P., Pozzati, G. & Elofsson, A. Nature Commun. 13, 1265 (2022).[10] Humphreys, I. R. et al. Science 374, eabm4805 (2021).
[11] Buel, G. R. & Walters, K. J. Nature Struct. Mol. Biol. 29, 1–2 (2022).
本文经授权转载自微信公众号“Nature Portfolio”,原文以What's next for AlphaFold and the AI protein-folding revolution为标题发表在2022年4月13日《自然》的新闻特写版块上。
0
推荐



 京公网安备 11010502034662号
京公网安备 11010502034662号 