阅读:0
听报道
编辑 | 药明康德内容团队
提到DeepMind公司,我们首先想到的可能是几年前,它开发的人工智能AlphaGo“横扫”顶尖人类围棋职业选手,变革了围棋的思考方式。除了在棋类比赛中所向披靡以外,DeepMind也在加速科学发现上迈出了重要一步。今日,DeepMind宣布,其新一代AlphaFold人工智能系统,在国际蛋白质结构预测竞赛(CASP)上击败了其余的参会选手,能够精确地基于氨基酸序列,预测蛋白质的3D结构。其准确性可以与使用冷冻电子显微镜(CryoEM)、核磁共振或 X 射线晶体学等实验技术解析的3D结构相媲美。这一突破被多家媒体称为“变革生物科学和生物医学”的突破。前基因泰克(Genentech)首席执行官Arthur D. Levinson博士称这一成就为“划时代的进步”(once in a generation advance)。
图片来源:DeepMind Blog
生物学50年来的重大挑战
我们都知道,蛋白质对生命来说是不可或缺的,它们支持生物体的几乎所有功能。这些复杂的大分子由氨基酸链构成,而蛋白质的功能很大程度上决定于它的3D结构。生物医学领域的众多挑战,包括开发治疗疾病的创新疗法,依赖于对蛋白质结构和功能的理解。
在过去的五十年中,科学家们已经能够利用冷冻电子显微镜、核磁共振或 X 射线晶体学等实验手段在实验室中确定蛋白质的形状,但每种方法都依赖于大量的试错,耗时耗力,可能需要花上好几年时间。1972年,诺贝尔化学奖得主Christian Anfinsen博士表示,理论上,蛋白质的氨基酸序列应该能够完全决定它的3D结构。这一假说激发了50年来基于氨基酸序列,通过计算方法预测蛋白质3D结构的探索。
然而,这一领域面临的重大挑战是理论上,氨基酸链可能形成的蛋白质构象的数目是个非常庞大的天文数字。有学者估计,一个典型的蛋白质理论上可以形成10的300次方(1后面加300个0)个可能构象。然而在自然界,蛋白质能够自发地在几毫秒内,迅速折叠成其中一个构象。用什么样的计算方法,才能从10的300次方的可能构象中找到那个正确的构象?
AlphaFold:生物界的“AlphaGo”
DeepMind的研究人员把折叠好的蛋白质设想成一幅具有3D结构的“空间图画”(spatial graph),而氨基酸则是这副“空间图画”中节点和线条。基于神经网络系统,他们设计了AlphaFold系统来解析这一空间图画的结构。它使用了进化相关的氨基酸序列,多序列对比(multiple sequence alignment, MSA)以及对氨基酸对(amino acid pairs)的评估来优化“空间图画“的描绘。

AlphaFold的神经网络模型构架丨图片来源:DeepMind Blog
研究人员使用蛋白质数据库中接近17万个不同的蛋白质结构,以及包含未知结构的蛋白序列数据库对AlphaFold进行训练。通过不断地迭代,AlphaFold系统学习到了基于氨基酸序列,精确预测蛋白结构的能力。
与实验结果相差无几的蛋白质结构预测
国际蛋白质结构预测竞赛(CASP)是由马里兰大学的John Moult教授和加州大学戴维斯分校的Krzysztof Fidelis教授联合创建的国际性比赛,旨在评估、促进和确认最佳的蛋白质结构预测手段。CASP选择已经通过实验手段解析,但是尚未公布的蛋白质结构作为目标,让世界各地的研究团队运用自己的计算手段预测它们的结构。一个独立的团队会评估预测结构与通过实验手段解析的蛋白结构之间的差异。
2018年,DeepMind开发的第一代AlphaFold首次参加CASP并且拔得头筹。而今年,新一代的AlphaFold在CASP中的表现更为惊艳。CASP使用称为GDT的评分系统来评估预测蛋白结构的精确性。这个评分从0到100,如果评分达到90分以上,可以认为预测的结构与实验手段获得的结构相当。
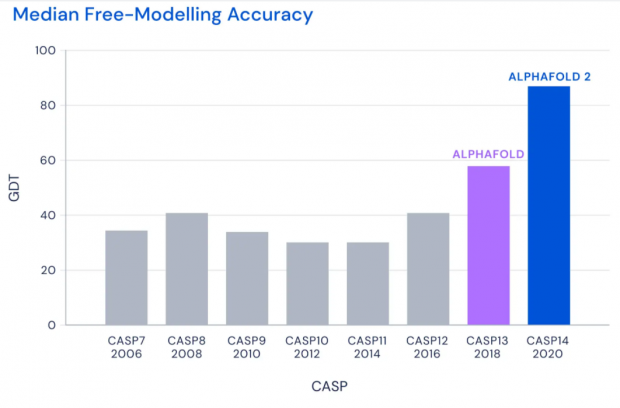
2006-2020年CASP比赛中最佳蛋白折叠预测系统的评分表现丨图片来源:DeepMind Blog
在今年的CASP中,AlphaFold系统对所有蛋白靶点3D结构预测的中位GDT评分为92.4分。即便是针对最难解析的蛋白靶点,AlphaFold的中位GDT评分也达到了87.0分。在接受检验的近100个蛋白靶点中,AlphaFold对三分之二的蛋白靶点给出的预测结构与实验手段获得的结构相差无几。CASP创始人Moult教授表示,在有些情况下,已经无法区分两者之间的区别是由于AlphaFold的预测出现错误,还是实验手段产生的假象。
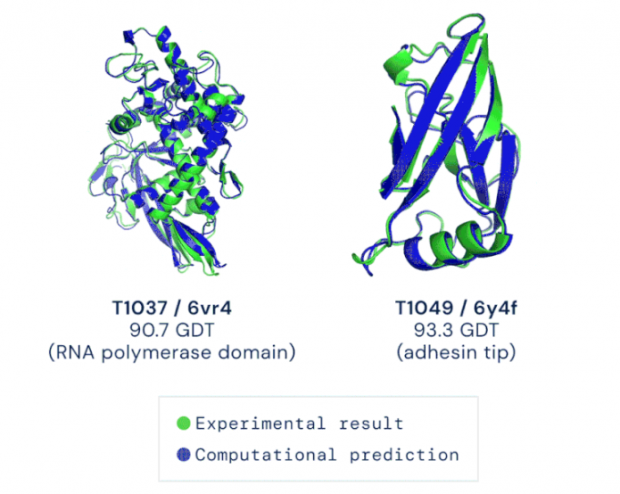
AlphaFold根据氨基酸序列预测的蛋白结构与实验手段解析的结果几乎完全重合丨绿色,实验结果;蓝色,计算预测结果;图片来源:DeepMind Blog
对真实世界的影响
在今年早些时候,DeepMind已经利用这一系统预测了多种新冠病毒蛋白的结构。后续的实验显示,AlphaFold预测的新冠病毒Orf3a蛋白结构与冷冻电镜解析的结构非常相似。
虽然,AlphaFold不见得会取代冷冻电子显微镜等其它实验手段,但是DeepMind的研究人员表示,这一令人兴奋的结果表明,生物学家们可以使用计算结构预测作为科学研究的核心工具之一。这一手段对于特定类型的蛋白来说可能尤为便利,例如膜蛋白一直非常难于结晶,因此很难用实验手段获得它们的结构。
而对于从事计算和机器学习研究的DeepMind团队来说,AlphaFold的表现证明了AI在辅助基础科学发现方面惊人的潜力。该团队在公司发布的博文中表示,他们相信,AI将成为人类拓展科学知识前沿最有力的工具之一!
参考资料
[1] AlphaFold: a solution to a 50-year-old grand challenge in biology. Retrieved November 30, 2020, from
[2] ‘It will change everything’: DeepMind’s AI makes gigantic leap in solving protein structures. Retrieved November 30, 2020, from
[3] CRITICAL ASSESSMENT OF TECHNIQUES FOR PROTEIN STRUCTURE PREDICTION. Retrieved November 30, 2020, from
本文经授权转载自微信公众号“药明康德”。
话题:
0
推荐
财新博客版权声明:财新博客所发布文章及图片之版权属博主本人及/或相关权利人所有,未经博主及/或相关权利人单独授权,任何网站、平面媒体不得予以转载。财新网对相关媒体的网站信息内容转载授权并不包括财新博客的文章及图片。博客文章均为作者个人观点,不代表财新网的立场和观点。




 京公网安备 11010502034662号
京公网安备 11010502034662号 